www.mundonano.unam.mx | Mundo Nano
http://dx.doi.org/10.22201/ceiich.24485691e.2022.29.69703 | 15(29), 1e-18e, julio–diciembre 2022
Artículos de investigación
TiO2 como nanoacarreadores de antibióticos (quinolonas): ensayo de acoplamiento molecular◊
TiO2 as nanocarriers of antibiotics (quinolones): A molecular docking assay
Esthela-Paola García-Tejada*
Faustino Aguilera-Granja**
Ángel Albino-Flores*
Adán Bazán-Jiménez***
Erik Diaz-Cervantes*♦
* Universidad de Guanajuato, Centro Interdisciplinario del Noreste (CINUG), Departamento de Alimentos.
** Universidad Autónoma de San Luis Potosí, Instituto de Física.
*** Universidad de Guanajuato, División de Ciencias Naturales y Exactas, Departamento de Química.
♦ Autor de correspondencia: e.diaz@ugto.mx
Resumen:
El objetivo del presente trabajo fue realizar un estudio QSAR considerando quinolonas comerciales para el desarrollo de nuevas estructuras químicas con posible acción antibiótica. Además, se complementó con un ensayo de acoplamiento molecular para conocer la interacción molecular entre las moléculas estudiadas y la ADN girasa-topoisomerasa IV (ADNg-TIV) de Staphylococcus aureus (S. aureus). Subsecuentemente, se realizó el acoplamiento molecular in silico de un modelo de TiO2 como vehículo de quinolonas. Todo esto fue desarrollado a partir de la teoría de funcionales de la densidad (DFT, por sus siglas en inglés), con el funcional M06L y un conjunto de base 6-31G(d,p); el docking molecular se realizó considerando la función de scoring MolDock Score. A partir de los resultados obtenidos en el estudio QSAR, se logró diseñar una familia de moléculas con valores de concentración inhibitoria mínima (MIC, por sus siglas en inglés) hasta mil veces menores que los fármacos comerciales; principalmente adicionando sustituyentes azoles e isatinas a los núcleos base. Los acoplamientos moleculares de nuestras moléculas diseñadas imitaron el mecanismo de acoplamiento con el blanco biológico como se ha reportado en la literatura. Estos resultados fueron aún más alentadores, mostrando energías más bajas y estables cuando las quinolonas interactúan con un nanoacarreador de TiO2. Finalmente, podemos concluir que obtuvimos cuatro moléculas con alto potencial debido a su MIC calculado, las cuales potencializan su acoplamiento molecular al utilizar un nanoacarreador de TiO2, siendo una propuesta sólida para futura síntesis y pruebas in vitro.
Palabras clave: nanoacarreador-TiO2, quinolonas, M06L/6-311+G (d,p), acoplamiento molecular, ADN-girasa, topoisomerasa IV.
Abstract:
A QSAR study was carried out, by considering common quinolone to develop new chemical structures with possible antibacterial action. Furthermore, it has been performed a docking assay to get insight into the molecular interactions of the studied molecules herein with the biological target Staphylococcus aureus (S. aureus). On the other hand, a TiO2 cluster was used as a nanocarrier model and was coupled to quinolones through a molecular docking assay. All the molecules were optimized using the density functional theory (DFT), with the functional M06L and a basis set 6-311+G(d,p); the molecular docking assay between isolated quinolone and the TiO2-quinolone complex with DNA gyrase-topoisomerase IV (like biological target), was performed with Molegro Virtual Docker Software. The results show that the designed molecules present MIC (minimum inhibitory concentrations) values better than those of commercial drugs, especially when isatine and azole substituents were added. The molecular coupling of the designed structures mimics the linkage mechanism with the biological target, as has been reported in the literature. The addition of TiO2 as a nanocarrier confers a better interaction than free drugs or free molecules due to the many target-ligand interactions. Finally, we can conclude that four potential antibiotics were designed, showing lower MIC values. Likewise, the TiO2 nanocarrier promotes a better molecular coupling with the biological target, being our molecules a solid proposal to be synthesized and in vitro tested.
Keywords: nanocarrier-TiO2, quinolones, M06L/6-311+G (d,p), molecular coupling, DNA-gyrase, topoisomerase IV.
Recibido: 29 de septiembre, 2021.
Aceptado: 19 de enero, 2022.
Introducción
En el área médica, las infecciones nosocomiales son de interés en salud pública por afectar entre el 5 y el 10% de pacientes que ingresan a un hospital (Mazón Sánchez y Verneiulle Román 2019), siendo factores de importancia la morbilidad y mortalidad en personas de edad avanzada, con enfermedades subyacentes, pacientes inmunosuprimidos o con enfermedades inflamatorias sistémicas (Maguiña Vargas, 2016). Se estiman 700 mil muertes al año, y se pronostica que la cifra ascenderá a 10 millones en 2050 (Oteo Iglesias, 2019). Estas infecciones se desarrollan debido a la resistencia de las cepas bacterianas a los antibióticos que actualmente se usan, por lo cual, la comunidad médica ha recurrido al uso de medicamentos más agresivos, o a mayor tiempo y/o mayores dosis de los mismos. Estas enfermedades en conjunto generan mayores gastos, convirtiéndose en un negocio mundial con inversiones económicas millonarias (Lebeque Pérez et al., 2006).
Una de las bacterias asociadas con infecciones nosocomiales es Staphylococcus aureus, la cual es una de las principales bacterias con resistencia bacteriana. Se le atribuye un mayor interés por los mecanismos de autoprotección y resistencia que ha generado, derivándose de ella una cepa resistente al antibiótico meticilina (S. aureus meticicilina-resistente = SAMR), lo cual limita las posibilidades farmacológicas de erradicar este tipo de infecciones (Cervantes-García et al., 2014; McGuinness et al, 2017). La resistencia se genera debido a la capacidad bacteriana de seguir replicándose aún en condiciones que normalmente son tóxicas o de destrucción para ellas (Oteo Iglesias, 2019). El SAMR muestra mutaciones en el gen gyrA de la cadena ADN girasa A y en el gen parE de la topoisomerasa IV (Jacoby, 2005; Tenover, 2006). Otro mecanismo de resistencia es a través de la permeabilidad de la membrana (Jacoby, 2005; Rodríguez-Martínez, 2005).
Los antibióticos se pueden clasificar por su espectro y mecanismo de acción (Seija y Vignoli, 2006). Algunos de los antibióticos con mecanismos de acción más efectivos son las quinolonas, por su inhibición en la transcripción y replicación del ADN de la bacteria, generando la intercalación del antibiótico entre el surco mayor de la cadena de ADN y la unión con la proteína topoisomerasa IV (indispensable para el proceso de replicación), lo cual interrumpe la secuencia del proceso e induce la muerte bacteriana (Alos, 2009; Rodríguez-Martínez, 2005; Campos, et al. 2008). La estructura química de las quinolonas consta de dos anillos aromáticos con un nitrógeno en la posición 1, un ácido carboxílico en el carbono 3 y un grupo carbonilo en el carbono 4 (Figura 1).
Figura 1 Estructura base de una quinolona. Número CAS 13721-01-2 (ácido 1,4-dihidro-4-oxo-3-quinolincaboxílico) consta de un doble anillo aromático con un átomo de nitrógeno en la posición uno, ácido carboxílico en la posición tres y un grupo carbonilo en la posición cuatro.
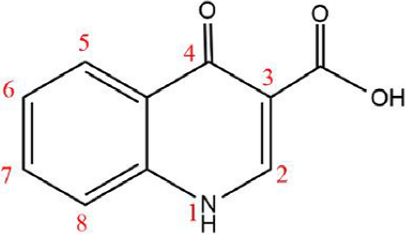
Fuente: Elaboración de los autores.
La evolución de las quinolonas se dio en primera instancia adicionando al núcleo base átomos de flúor (fluoroquinolonas), átomos de cloro (clinafloxacina), grupos amino (esparfloxacina), hidroxilos, difluorofenilo (trovafloxacina) y éter difluorometilico (garenoxacina) (Pham, Ziora, y Blaskovich, 2019). La resistencia bacteriana se puede evitar si se alcanza y mantiene una concentración sérica del fármaco en el cuerpo, para el suministro adecuado intracelularmente (Cué Brugueras, Morejón García y Salup Díaz, 2005; Seija y Vignoli, 2006).
Dentro de la tecnología farmacéutica se han ideado nuevos métodos para la entrega eficiente, inteligente y selectiva de fármacos, lo cual permite una dosificación necesaria evitando reacciones secundarias o daños adversos. El uso de nanoacarreadores de TiO2 es una alternativa para la entrega y liberación del fármaco al blanco biológico. Actualmente, se ha utilizado este tipo de nanopartículas como vehículo de fármacos contra el cáncer, siendo un éxito al extender su dosificación en las células cancerígenas, induciendo la muerte celular selectiva y minimizando los efectos secundarios (T. Wang et al., 2015; Q. Wang et al., 2016).
De igual manera, el uso de la química computacional nos permite diseñar nuevos fármacos por medio de estudios cuantitativos estructura-actividad (QSAR, por sus siglas en inglés), así como conocer y predecir la eficiencia y el comportamiento de los posibles fármacos con algún blanco biológico, por medio de acoplamientos moleculares in silico (Pérez, 1994; Bitencourt-Ferreira y de Azevedo, 2019; De Azevedo, 2019; Lengauer y Rarey, 1996; Lozano-Aponte y Scior, 2012; Rogers y Hopfinger, 1994).
Considerando lo anterior, en el presente trabajo se realizó el modelado y estudio computacional de una familia de quinolonas comerciales, con el objetivo de diseñar nuevos compuestos con actividad antibiótica. Lo anterior comenzando con un estudio QSAR de los fármacos comerciales, para conocer la actividad que confiere la estructura de las mismas. El siguiente paso fue diseñar nuevas moléculas a partir de los datos generados por el QSAR, para, posteriormente, evaluar las interacciones de las quinolonas comerciales y diseñadas con un blanco presente en S. aureus, a partir de acoplamientos moleculares in silico. Por último, a partir de un modelo finito de TiO2, se planteó la interacción de dicho modelo con las quinolanas estudiadas, para evaluar el efecto de la adición de dicho compuesto (simulando un nanoacarreador), al interactuar con el blanco biológico de S. aureus.
Métodos computacionales
Los resultados que se obtuvieron en el presente trabajo, empleando la infraestructura del Laboratorio de Caracterización Molecular de Biosistemas y Nanocompuestos (LACAMBION),1 se realizaron, gracias a ello, en un tiempo record, y mediante una infraestructura de alto rendimiento (denominado “La Biznaga”) se llevaron a cabo masivamente cálculos numéricos distribuyendo las operaciones/cálculos en dos nodos dedicados a alto rendimiento y disponibilidad, con un almacenamiento redundante de 16 terabytes y balanceando la carga en un nodo de acceso.
Optimizacion de las quinolonas comerciales y el nanoacarreador
Se realizó el modelado y la optimización del nanoacarreador de TiO2 , así como de una familia de fármacos comerciales de diversas generaciones (segunda a cuarta generación), tales como el ácido oxolínico (Oxo), ciprofloxacina (Cipro), ofloxacina (Oflo), enoxacina (Eno), norfloxacina (Nor), lomefloxacina (Lom), levofloxacina (Lev), esparfloxacina (Esp), clinafloxacina (Clin), gatifloxacina (Gat), trovafloxacina (Tro) y moxifloxacina (Mox) (Figura 2). Cabe destacar que estos fármacos fueron seleccionados por ser utilizados en el área clínica. El modelado fue realizado por medio del software Avogadro, posteriormente se realizó la optimización y el cálculo de descriptores electrónicos empleando la teoría del funcional de la densidad (DFT) mediante el uso del paquete computacional Gaussian 09, y, específicamente, aplicando un nivel M06L/6-311+G(d,p).
Figura 2 Moléculas de quinolonas comerciales.

Fuente: Elaboración de los autores.
Diseño y optimizacion de quinolonas prospecto
Se diseñaron moléculas de núcleo quinolónico con adición y evaluación de diferentes grupos funcionales. Se realizaron cuatro categorías, a la primera se le adicionaron ciclos insaturados de tres, cuatro y seis carbonos; a este último se le adicionaron átomos de flúor para mejorar la distribución de la nube electrónica. La segunda y tercera categoría consistió en la adición de triazoles y tetrazoles, respectivamente, variando la posición de los átomos de nitrógeno y las saturaciones presentadas. Finalmente, a la cuarta categoría se le adicionaron isatinas con distintos puntos de anclaje a la quinolona (Figura 3). Las quinolonas prospecto fueron modeladas y optimizadas con la metodología descrita anteriormente.
Figura 3 Moléculas de quinolonas propuestas.

Nota: El diseño de quinolonas se clasificó en cuatro grupos, del grupo naranja se evaluaron distintos ciclos insaturados. Al grupo azul se le adicionaron triazoles con distintas saturaciones y nitrógenos en múltiples posiciones; al grupo verde se le adicionaron tetrazoles, con distintas saturaciones y con distinto acomodo de los nitrógenos. Por último, al grupo morado se le adicionaron isatinas como sustituyentes con distintos puntos de anclaje al núcleo de quinolona, el primero con unión en el nitrógeno y el segundo con unión al oxígeno próximo al nitrógeno.
Fuente: Elaboración de los autores.
Cálculo de descriptores electrónicos
A partir de la DFT conceptual, se calcularon los siguientes descriptores de reactividad global: potencial de ionización (I), afinidad electrónica (A), electronegatividad (χ), dureza (η), blandura (S), y, electrofilicidad (ω), los cuales fueron calculados mediante las ecuaciones mostradas en [1] (Robles et al., 2000).

Estudio QSAR
Para las estructuras de quinolonas comerciales se realizó un estudio QSAR empleando el software Dragon (Thiemann y Vill, 1997), a partir del cual se obtuvieron más de 1,200 descriptores geométricos, topológicos y moleculares. Posteriormente, se planteó un modelo matemático con el uso de los descriptores geométricos y electrónicos, para poder predecir el valor experimental MIC (Andersson y MacGowan, 2003; Hoogkamp-Korstanje, 1997; Prosser y Beskid, 1995; Pham, Ziora y Blaskovich, 2019; Yoshimura, Ishimaru y Kojima, 2002). Para facilitar el manejo de datos se utilizó el pMIC definido en [2].
El modelo matemático predictivo o QSAR se obtuvo a partir del software MobyDigs (Todeschini et al., 2004).
Acoplamientos moleculares in silico
Se obtuvo la estructura cristalizada de la ADN girasa-topoisomerasa IV (ADNg-TIV) como blanco biológico del Protein Data Bank (código PDB: 5CDP) (Figura 4), lo anterior por ser un blanco biológico específico para derivados quinolónicos, sin considerar posibles mutaciones derivadas de la resistencia a antibióticos. Se evaluó la interacción entre las quinolonas y el composito de quinolona-TiO2 en las cavidades del blanco biológico, por medio del algoritmo de búsqueda MolDock SE y una función de evaluación MolDock Score (GRID) (Thomsen y Christensen, 2006). Con lo anterior se lograron conocer las poses energéticamente más estables y la energía de interacción blanco-ligando. El análisis se realizó con el uso del software Molegro Virtual Docker (Thomsen et al., 2006).
Figura 4 ADNg-TIV como blanco biológico de quinolonas en un modelo de S. aureus.
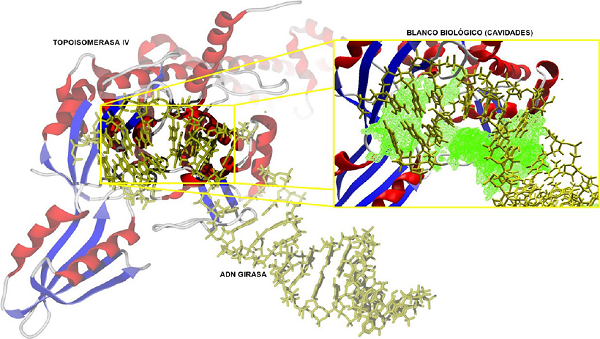
Nota: Las cavidades (verde) identifican regiones con probabilidad de interacciones.
Fuente: Elaboración de los autores.
Resultados y discusión
Modelo QSAR
Los descriptores obtenidos fueron del tipo topológicos, geométricos, de cargas, grupos funcionales, atómicos y de propiedades moleculares, los cuales se evaluaron en los distintos modelos matemáticos desarrollados mediante algoritmos evolutivos. En la selección del modelo matemático óptimo, se evaluaron distintos elementos; el primero de ellos es que tuviera un coeficiente de correlación (R2) cercano a uno, además de un análisis de componentes principales (PC), el cual está condicionado a que los descriptores seleccionados dentro del modelo matemático no tengan alguna correlación lineal entre ellos, pero sí con el pMIC como valores responsivos de la generación del modelo matemático. Previamente se validó el modelo con un conjunto de prueba aleatorio. Los valores obtenidos en el modelo seleccionado presentaron un valor de R2 = 0.97, y en el análisis PC resultó SCBO ≠ ARR ≠ G(N..O), por lo que estos descriptores no tienen alguna correlación entre ellos, pero sí con PMIC (Gráfica 1). El modelo matemático obtenido se muestra en [3].

Gráfica 1 Comparación de pMIC experimental contra pMIC in silico.
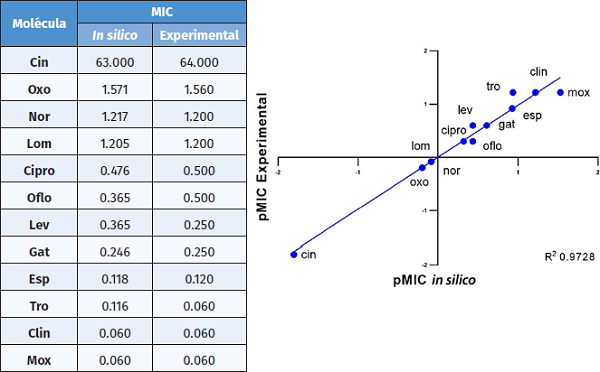
Fuente: Elaboración de los autores.
Donde SCBO es el orden de suma en enlaces convencionales para el modelo matemático en estudio (MME); ARR indica el radio aromático; MAATS3v es la autocorrelación de Moran en volúmenes de Van Der Waals, y G(N..O) es la suma de distancias geométricas entre los átomos de nitrógeno y oxígeno.
Para el diseño de nuevas quinolonas se consideraron los sustituyentes con mayor impacto en la eficiencia del fármaco, reportados en la bibliografía y en estudios previos de nuestro grupo de investigación. Se conservó el grupo metoxilo en el sexto carbono, la piperazina en el séptimo carbono y, finalmente, el flúor en el octavo carbono, considerando esta base como nuestro andamio para nuevas propuestas. Al evaluar estas estructuras nuevas con el MME y los descriptores, podremos conocer el valor teórico de pMIC y por tanto de MIC (Tabla 1), teniendo así una referencia de la posible inhibición de S. aureus por estas estructuras.
Tabla 1 Valores teóricos de pMIC y MIC para las estructuras quinolónicas propuestas.
* pMIC: Es una normalización logarítmica de MIC para estandarizar los valores experimentales, la manera de interpretación: el valor pMIC > mejor efectividad del antibiótico. ** MIC: Concentración mínima inhibitoria, por lo que mientras MIC < cantidad en mg/L se requiera del fármaco para inhibir una cepa bacteriana, mejo efectividad del antibiótico. Nota: Las moléculas 1-4, 16 y 17 tienen valores dentro del rango de las quinolonas comerciales. Las moléculas 5-15, muestran valores hasta mil veces por debajo de las quinolonas comerciales. Fuente: Elaboración de los autores.
Molécula
In silico
pMIC*
pMIC**
1
-0.094
1.24229
2
0.269
0.53798
3
0.769
0.17025
4
1.740
0.01820
5
3.250
0.00056
6
2.701
0.00199
7
4.389
0.00004
8
3.517
0.00030
9
3.645
0.00023
10
3.778
0.00017
11
4.407
0.00004
12
4.086
0.00008
13
3.519
0.00030
14
3.630
0.00023
15
3.777
0.00017
16
0.593
0.25530
17
0.982
0.10419
A través de las estructuras iniciales, se logró conocer el efecto del sustituyente 2,4-difluorofenilo, en comparación con el resto de las estructuras con anillos insaturados; obteniendo un pMIC = 1.74, el cual, en comparación con el pMIC = 1.22 de Mox, Clin y Tro (quinolonas comerciales de mayor eficiencia), es mayor, lo cual indica un mejor efecto en la propuesta. En cuanto a las moléculas 16 y 17 con un grupo isatina, se presenta un pMIC de 0.593 y 0.982, respectivamente, ubicándose en el promedio de las quinolonas comerciales.
En cuanto al resto de las moléculas con sustituyentes triazoles y tetrazoles supera hasta en mil unidades los pMIC experimentales, en especial las moléculas 11 y 12 en el grupo de tetrazoles y la molécula 7 en la clasificación de triazoles 4.407, 4.086 y 4.389, respectivamente. Una característica de estas últimas estructuras es la presencia de nitrógenos en las posiciones 1, 2, 6 del azol unido al nitrógeno en la primera posición de la base quinolónica. Dado lo cual, en esta región concentrada de nitrógenos, en conjunto con el flúor próximo, generan una densidad electrónica localizada, mayor electronegatividad, y por lo tanto presentan un comportamiento de nucleófilo.
Una diferencia entre la estructura 7 con la 11 y 12, es que la primera solo tiene tres nitrógenos pertenecientes al anillo, mientras que las otras estructuras presentan cuatro dentro del anillo, esto sin considerar el nitrógeno al que se encuentran unidos. Al parecer, el contener solo tres nitrógenos es mejor que contener cuatro en el anillo sustituyente, para obtener valores mayores en pMIC (Gráfica 2). En las estructuras 9, 10, 14 y 15, se evaluaron los dobles enlaces en los azoles, indicando un aporte positivo al presentar dos insaturaciones en lugar de solo una.
Gráfica 2 MIC in silico de quinolonas propuestas.
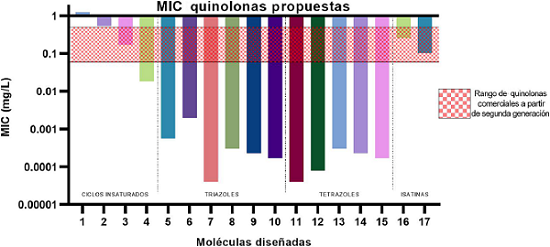
Nota: El valor expresado en MIC nos indica la cantidad necesaria para inhibir el crecimiento de una cepa bacteriana, lo deseable dentro del área farmacéutica e investigación es que sea una mínima cantidad para que tenga efecto, en otras palabras, mientras más pequeño sea el valor MIC se estipula una mejor quinolona.
Fuente: Elaboración de los autores.
Los valores MIC obtenidos in silico para las quinolonas propuestas con triazoles, tetrazoles en diferentes saturaciones e isatinas con diferentes puntos de anclaje, se encuentran hasta mil unidades por debajo de los MIC comerciales (sugiriéndose una eficiencia 1,000 veces mejor). Por lo tanto, de acuerdo con el estudio QSAR, las moléculas incluidas en este estudio se pueden considerar como prometedoras; sin embargo, esto no es evidencia suficiente. Es por ello que se realizó un ensayo de acoplamiento molecular, con el objetivo de conocer las interacciones que cada estructura exhibe en contacto con su blanco biológico.
Acoplamientos moleculares in silico
Quinolonas comerciales y diseñadas
Se realizaron acoplamientos moleculares de las quinolonas comerciales y diseñadas, resultando un acoplamiento en el surco mayor de la cadena de ADNg por la alta afinidad presentada. Cabe destacar que entre más negativo sea el valor de energía de acoplamiento ligando-receptor, el sistema se torna más estable. Esto ocurrió en todos los casos, comprobando el mecanismo de inhibición presentada en la literatura. Las estructuras con mejores resultados de MIC fueron: Tro con una energía de acoplamiento de -152.90 kcal/mol, Clin con -147.52 kcal/mol, Mox con -146.89Kcal/mol, y, Esp con -137.35Kcal/ mol en el caso de las comerciales.
Las interacciones presentes son de tipo puente de hidrógeno (PH) en las cuatro estructuras con los anillos y grupos fosfato del ADNg. No obstante, Tro y Mox también presentaron interacciones estéricas (IE) con la proteína topoisomerasa IV (pTIV) en los aminoácidos serina-glutamina y serina-glicina, respectivamente (Tabla 2). Otra interacción observada en las quinolonas es la interacción tipo apilamiento (overlaping o staking), esta última simula un emparedado donde el fármaco se encuentra atrapado por los anillos de las bases nitrogenadas de la cadena.
Tabla 2 Energías de acoplamiento en quinolonas comerciales y diseñadas.
Aminoácidos en interacción: serina (Ser), glutamina (Glu), glicina (Gly), aspártico (Asp). El número seguido indica la posición del aminoácido en la secuencia en la proteína. Nota: Se muestran las interacciones en azul para PH y en naranja para IE de cada molécula. Fuente: Elaboración de los autores.
Molécula
Energía de
Interacciones*
Cin
-119.709
ADNg
Oxo
-117.743
Nor
-130.891
ADNg, Ser 84
Lom
-129.941
Cipro
-134.091
ADNg
Oflo
-134.667
Lev
-136.444
Gat
-136.743
ADNg, Ser 84
Esp
-137.357
ADNg
Tro
-152.905
ADNg, Glu
88,
Clin
-147.525
ADNg
Mox
-146.899
ADNg, Gly
82,
Molécula
Energía de
Interacciones
1
-127.570
ADNg, Ser 84
2
-133.541
ADNg
3
-144.750
ADNg, Ser 85
4
-133.842
ADNg
5
-140.478
ADNg, Ser 85
6
-136.898
ADNg
7
-129.163
ADNg, Ser 85
8
-138.870
9
-142.473
ADNg
10
-137.091
11
-135.840
12
-134.970
13
-135.801
14
-136.728
15
-140.234
16
-152.326
ADNg, Ser
85,
17
-146.899
ADNg, Ser
85,
En cuanto a las quinolonas diseñadas, el acoplamiento también ocurrió en el surco mayor del ADNg, obteniendo energías de acoplamiento comparables y en algunos casos más bajas que las de las quinolonas ya analizadas. Las quinolonas diseñadas, 9 y 3, presentaron energías de acoplamiento de -142.47 kcal/ mol y -142.47 kcal/mol, respectivamente, comparables a las mejores quinolonas comerciales Clin (-147.52 cal/mol) y Mox (-146.89 kcal/mol. Sin embargo, las estructuras 16 y 17 alcanzaron energías de acoplamiento menores, de -152.32 y -164.54, respectivamente (Tabla 2); en estas últimas estructuras también se encontró una mayor cantidad de interacciones del tipo PH e IE con los aminoácidos serina, asparagina y glutamina (Figura 5).
Figura 5 Acoplamientos moleculares de quinolonas comerciales (fondo blanco). Se muestra el acomodo que obtuvieron las quinolonas comerciales en el surco mayor del ADNg. Las quinolonas diseñadas se acoplaron de manera similar a las comerciales. Átomos interactuantes de quinolonas (fondo negro). Se muestran los átomos que se encuentran en interacción con el blanco biológico de las quinolonas comerciales y diseñadas, así como los distintos tipos de interacción: puentes de hidrógeno (línea punteada azul) en interacción con ADNg, interacciones estéricas (línea punteada roja) con pTIV y, finalmente, las interacciones tipo apilamiento (círculos verdes) con los anillos de las bases nitrogenadas de las cadenas.
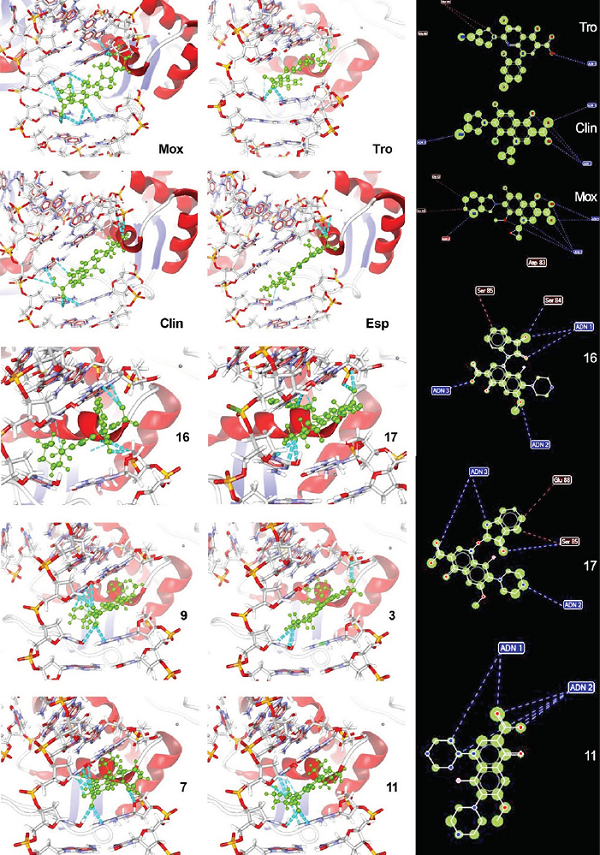
Nota: El valor expresado en MIC nos indica la cantidad necesaria para inhibir el crecimiento de una cepa bacteriana, lo deseable dentro del área farmacéutica e investigación es que sea una mínima cantidad para que tenga efecto, en otras palabras, mientras más pequeño sea el valor MIC se estipula una mejor quinolona.
Fuente: Elaboración de los autores.
Por otra parte, las estructuras 7 y 11 resultaron tener el MIC más alto en el modelo QSAR, obteniendo una energía de acoplamiento de -129.16Kcal/ mol y -135.84Kcal/mol, en ese orden y aunque en este análisis no exhibieron los mejores resultados, presentan interacciones con el ADNg con PH y apilamiento, mientras que la IE en el pTIV solo ocurrió con la estructura 7.
La correlación entre los resultados obtenidos del QSAR y los acoplamientos moleculares in silico se pueden explicar en términos farmacofóricos, considerando la estructura electrónica generada en los sistemas con mejor MIC.
Compósito de TiO2 con quinolonas comerciales y diseñadas
Se obtuvo la estructura optimizada del compósito de TiO2, resultando un mínimo en la superficie de energía potencial (Figura 6), la cual es un modelo finito de una nanopartícula de TiO2, obtenida de una celda unitaria de brookita con un tamaño de aproximádamente 7 Å, la cual fue pasivada con átomos de hidrógeno para evitar valencias libres. Se analizaron distintas interacciones con ADNg-TIV, para conocer la estabilidad de los ligandos, cuando se coordinan con dichos blancos. La energía de acoplamiento obtenida de la ADNg-TiO2 resultó de -67.43 kcal/mol. Los acoplamientos moleculares se realizaron primeramente entre el TiO2 con las quinolonas y los resultados mostraron energías de unión similares entre las quinolonas comerciales y las diseñadas.
Figura 6 Modelo optimizado de TiO2.

Nota: Las esferas grises denotan los átomos de titanio, las esferas blancas representan átomos de hidrógeno y las esferas rojas denotan átomos de oxígeno.
Fuente: Elaboración de los autores.
Posteriormente se realizó un acoplamiento del compósito con el blanco biológico mostrando dos regiones de posicionamiento, la primera se llevó a cabo a un costado de la cadena de ADNg mostrando la energía más estable, mientras que la segunda posición se desarrolló en el surco mayor como lo muestran las conformaciones anteriores con quinolonas libres.
Al realizar un acoplamiento de la quinolona con el nanoacarreador, se obtiene una energía más baja e incluso una mayor formación de interacciones intramoleculares formando PH con los nucleótidos del ADNg y los aminoácidos del pTIV (Gráfica 3). Lo anterior denota que es favorable el uso de un nanoacarreador para la entrega del fármaco, coadyuvando en la inhibición de la replicación del ADN bacteriano.
Gráfica 3 Comparación de energías de acoplamiento entre quinolonas y compósitos con TiO2.

Nota: Las energías de los compósitos de TiO2-quinolona resultaron ser más estables energéticamente, debido a los valores obtenidos al interactuar el ligando con el blanco biológico.
Fuente: Elaboración de los autores.
De igual manera, se realizó el análisis de las mejores quinolonas comerciales y diseñadas. Las quinolonas Mox, Clin, 7, 11, 16 y 17 mostraron la mejor energía de acoplamiento por un costado de la cadena de ADNg, mientras que en Tro y Esp mostraron una mejor energía de acoplamiento en la región del surco de la cadena (Tabla 3).
Tabla 3 Energías de acoplamiento de quinolonas y compósitos, con el blanco seleccionado.
* Aminoácidos en interacción: serina (Ser), glutamina (Glu), glicina (Gly), aspártico (Asp), arginina (Arg), valina (Val), metionina (Met), asparangina (Asn), tirosina (Tyr). El número seguido indica la posición del aminoácido en la secuencia en la proteína. ** Pose 1: Acoplamiento molecular en un costado de la cadena de ADNg. *** Pose 2: Acoplamiento molecular en el surco mayor de la cadena de ADNg. Fuente: Elaboración de los autores.
Energías de acoplamiento molecular (kcal/mol)
Molécula
ADNg-
ADNg-TiO2-
Interacciones*
Pose 1**
Pose 2***
Mox
-146.899
-165.486
-
ADNg, Glu88, Ser85, Arg92
Clin
-147.525
-155.327
-
ADNg, Val268, Met113, Asn269, Gly115, Arg92, Glu88
Tro
-155.741
-
-168.697
ADNg, Glu88, Ser84
Esp
-137.357
-
-145.119
ADNg, Glu88, Ser84
7
-129.163
-141.113
-
ADNg, Glu88, Gln95, Arg92
11
-135.840
-139.021
-
ANDg, Ala118, Val91, Arg92-272, Gly117
16
-152.326
-161.167
-
ADNg, Ser84, Tyr87, Ala118
17
-164.548
-169.168
-
ADNg, Ala120, Tyr87, Glu88, Ser84
Las estructuras con mejor energía de acoplamiento fueron las 17, 16, 7 y 11 con -169.168 kcal/mol, -161.167 kcal/mol, -141.113 kcal/mol y -139.021 kcal/mol, respectivamente. En los casos de las quinolonas diseñadas se obtuvieron al menos cuatro interacciones con los átomos de fosforo, oxígeno y nitrógeno de la cadena de ADNg y los aminoácidos de pTIV (Figura 7). La formación de estas interacciones intermoleculares permite tener un mayor anclaje al blanco biológico.
Figura 7 Interacciones de los compósitos de TiO2-quinolona con ADNg-TIV.

Nota: Se muestran las interacciones PH (líneas punteadas en azul) e IE (líneas punteadas en naranja) con los diferentes aminoácidos de la proteína y con los distintos átomos pertenecientes a la cadena.
Fuente: Elaboración de los autores.
Conclusiones
Los estudios in silico nos permiten estudiar los mecanismos moleculares que existen para una serie de fármacos, regulando una gran cantidad de recursos económicos, consumibles y de tiempo efectivo. Haciendo uso de estudios QSAR fue posible obtener valores teóricos del MIC para un grupo de moléculas diseñadas, resultando valores favorables, en conjunto con acoplamientos computacionales. Lo anterior nos permitió conocer las regiones con mayor afinidad de unión en el blanco biológico.
Las estructuras 7 y 11 mostraron valores de MIC mil veces mejores que los fármacos comerciales. Sin embargo, no se obtuvieron los valores más bajos en el acoplamiento molecular, quedando en un valor promedio con respecto a los valores obtenidos en las quinolonas comerciales; cabe destacar que las diferencias de valores no son significativas y están en el mismo orden de magnitud. Estos valores de acoplamiento se vieron favorecidos al vincularse con el nanoacarreador de TiO2. Las quinolonas diseñadas 16 y 17 mostraron un MIC en el promedio de las quinolonas comerciales de acuerdo con el estudio QSAR. En cuanto a la energía de acoplamiento, quedó claro que el uso de un nanoacarreador de de TiO2 promueve una mejor interacción blanco-ligando, a través de interacciones de puente de hidrógeno, electrostáticas y de apilamiento. De acuerdo con los resultados, los grupos funcionales adicionados generaron mejores valores de MIC al adicionar azoles; no obstante, en las interacciones de acoplamiento molecular y sus energías, el uso de isatinas resultó más favorable, en concordancia con lo reportado en la literatura científica, indicando su eficiencia como antibióticos. Por lo anterior, las moléculas diseñadas y reportadas en el presente trabajo se consideran modelos prometedores para un estudio in vitro.
El uso de nanoacarreadores es un amplio campo de estudio promisorio para el éxito en la entrega inteligente, moderada y objetiva de fármacos en el campo médico, lo cual queda demostrado en este trabajo de investigación. A partir del presente estudio fue posible generar una nueva familia de antibióticos-nanoestructurados, que pueden ser considerados candidatos a fármacos.
Referencias
- Alos, Juan-Ignacio. 2009. Quinolonas. Enfermedades infecciosas y microbiología clínica, 27(5): 290-297.
- Andersson, Monique I. y Alasdair P MacGowan. 2003. Development of the quinolones. Journal of Antimicrobial Chemotherapy, 51 (suppl_1): 1-11. https://doi.org/10.1093/jac/dkg212
- Bitencourt-Ferreira, Gabriela y Walter Filgueira de Azevedo. 2019. Molegro virtual docker for docking. Docking Screens for Drug Discovery, 149-167. Springer Nature Experiments.
- Campos, S., Alfonso E., Martínez Enríquez, María E., Mendoza Patiño, N. 2008. Quinolonas. Revista de la Facultad de Medicina UNAM, 51(4): 173-177.
- Cervantes-García, Estrella, Rafael García-González y Paz María Salazar-Schettino. 2014. Características generales del Staphylococcus aureus. Revista Mexicana de Patología Clínica y Medicina de Laboratorio, 61(1): 28-40.
- Cué Brugueras, Manuel, Moisés Morejón García y Rosa Salup Díaz. 2005. Actualidad de las quinolonas. Revista Cubana de Farmacia, 39(1): 1-1.
- De Azevedo, Walter Filgueira. 2019. Docking screens for drug discovery. Springer.
- Hoogkamp-Korstanje, J. A. 1997. In vitro activities of ciprofloxacin, levofloxacin, lomefloxacin, ofloxacin, pefloxacin, sparfloxacin and trovafloxacin against gram-positive and gram-negative pathogens from respiratory tract infections. The Journal of antimicrobial chemotherapy, 40(3): 427-431. https://doi.org/10.1093/jac/40.3.427
- Jacoby, George A. 2005. Mechanisms of resistance to quinolones. Clinical infectious diseases, 41 (Suppl. t_2): S120-S126. https://doi.org/10.1086/428052
- Lebeque Pérez, Yamila, Humberto J. Morris Quevedo y Nerys Calás Viamonte. 2006. Infecciones nosocomiales: incidencia de la Pseudomonas aeruginosa. Revista Cubana de Medicina, 45(1).
- Lengauer, Thomas y Matthias Rarey. 1996. Computational methods for biomolecular docking. Current opinion in structural biology 6(3): 402-406. https://doi.org/10.1016/S0959-440X(96)80061-3
- Lozano-Aponte, Jorge Thomas Scior. 2012. ¿Qué sabe Ud. acerca de... QSAR? Revista Mexicana de Ciencias Farmacéuticas, 43(2): 82-84. https://www.redalyc.org/articulo.oa?
- Maguiña Vargas, Ciro. 2016. Infecciones nosocomiales. Acta Médica Peruana, 33(3): 175-177.
- Mazón Sánchez, Jenniffer Anabel y Jorge Stanislao Verneiulle Román. 2019. Factores de riesgos que influyen en el desarrollo de las enfermedades nosocomiales. Repositorio Dspace. http://doi.org/repositorio.unemi.edu.ec/handle/123456789/4709
- McGuinness, Will A., Natalia Malachowa y Frank R. DeLeo. 2017. Vancomycin resistance in Staphylococcus aureus. The Yale Journal of Biology and Medicine, 90(2): 269.
- Oteo Iglesias, Jesús. 2019. Comprendiendo la resistencia a antibióticos. Revista de Investigación y Educación en Ciencias de la Salud, 4(2). https://doi.org/10.37536/RIECS.2019.4.2.164
- Pham, Thu D. M., Zyta M. Ziora y Mark A. T. Blaskovich. 2019. Quinolone antibiotics. MedChemComm, 10(10): 1719-1739.
- Prosser, Barbara La T. y George Beskid. 1995. Multicenter in vitro comparative study of fluoroquinolones against 25,129 gram-positive and gram-negative clinical isolates. Diagnostic Microbiology and Infectious Disease, 21(1): 33-45. https://doi.org/10.1016/0732-8893(94)00087-D
- Pérez, María Selma Arias. 1994. Mecánica molecular. Diseño de medicamentos, Dialnet, España
- Robles, J., J. Martin-Polo, L. Alvarez-Valtierra, L. Hinojosa y G. Mendoza-Díaz. 2000. A theoretical-experimental study on the structure and activity of certain quinolones and the interaction of their Cu (II)-complexes on a DNA model. Metal-Based Drugs, 7(6): 301. https://doi.org/10.1155/MBD.2000.301
- Rodríguez-Martínez, José Manuel. 2005. Mecanismos de resistencia a quinolonas mediadas por plásmidos. Enfermedades Infecciosas y Microbiología Clínica, 23(1): 25-31. Elsevier. https://doi.org10.1157/13070406
- Rogers, David y Anton J Hopfinger. 1994. Application of genetic function approximation to quantitative structure-activity relationships and quantitative structure-property relationships. Journal of Chemical Information and Computer Sciences, 34(4): 854-866. https://doi.org/10.1021/ci00020a020
- Seija, V. y R Vignoli. 2006. Principales grupos de antibióticos. Temas de Bacteriología y Virología Médica, 2: 631-633.
- Tenover, Fred C. 2006. Mechanisms of antimicrobial resistance in bacteria. American Journal of Medicine, 119(6): S3-S10. https://doi.org/10.1016/j.amjmed.2006.03.011
- Thiemann, Thies y Volkmar Vill. 1997. Preliminary communication: Development of an incremental system for the prediction of the nematic-isotropic phase transition temperature of liquid crystals with two aromatic rings. Liquid Crystals, 22(4): 519-523. https://doi.org/10.1080/026782997209252
- Thomsen, René y Mikael H. Christensen. 2006. MolDock: a new technique for high-accuracy molecular docking. Journal of Medicinal Chemistry, 49(11): 3315-3321. https://doi.org/10.1021/jm051197e
- Todeschini, Ballabio D., Consonni V., Maui A. y Pavan M., 2004. “(2004) MobyDigs Computer Software-Version 1.0 2004 copyright TELETE srl.”.
- Wang, Qun, Jian-Ying Huang, Hua-Qiong Li, Allan Zi-Jian Zhao, Yi Wang, Ke-Qin Zhang, Hong-Tao Sun y Yue-Kun Lai. 2016. Recent advances on smart TiO(2) nanotube platforms for sustainable drug delivery applications. International Journal of Nanomedicine, 12: 151-165. https://doi.org/10.2147/IJN.S117498
- Wang, Tianyi, Haitao Jiang, Long Wan, Qinfu Zhao, Tongying Jiang, Bing Wang y Siling Wang. 2015. Potential application of functional porous TiO2 nanoparticles in light-controlled drug release and targeted drug delivery. Acta Biomaterialia, 13: 354-363. https://doi.org/10.1016/j.actbio.2014.11.010
- Yoshimura, H., M. Ishimaru y A. Kojima. 2002. Minimum inhibitory concentrations of 20 antimicrobial agents against Staphylococcus aureus isolated from bovine intramammary infections in Japan. Journal of Veterinary Medicine, Series B 49(9): 457-460. https://doi.org/10.1046/j.1439-0450.2002.t01-1-00593.x